Usługowe
genotypowanie mniejszościowych wariantów genetycznych
w niejednorodnym materiale biologicznym i klinicznym, z czułością 0,1% całkowitego DNA/RNA
Usługowe
genotypowanie mniejszościowych wariantów genetycznych
w niejednorodnym materiale biologicznym i klinicznym, z czułością 0,1% całkowitego DNA/RNA
Analiza podłoża genetycznego w niejednorodnym materiale biologicznym lub klinicznym (który może zawierać kilka różnych wariantów tego samego genu) wymaga zastosowania odpowiednich procedur analitycznych lub stosownej procedury oceny uzyskiwanych wyników przy zastosowaniu klasycznych metod genotypowania.
Problem niejednorodności materiału wyjściowego w analizie genetycznej jest obecny np. w onkologii przy analizie mutacji somatycznych w materiale z guza, lub w wirusologii, zwłaszcza w przypadku wirusów RNA, gdzie obecność kilku wariantów genetycznych wirusa w tym samym organizmie i tym samym czasie jest naturalnym zjawiskiem. Podobnie kilka wariantów genetycznych tego samego organizmu można się spodziewać w próbach mikrobiologicznych od pacjentów lub w próbach środowiskowych.
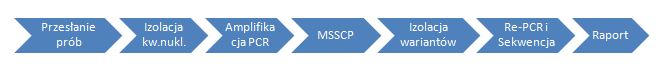
Oferujemy unikalną Usługę Genotypowania Niejednorodnego Materiału (PMES) biologicznego w oparciu o wynalezioną i opatentowaną przez nas metodę MSSCP (Multitemperature Single Strand Conformation Polimorphism). Procedura ta pozwala na detekcję znanych mniejszościowych wariantów genetycznych oraz na odkrycie zupełnie nowych wariantów, obecnych w badanej próbie na poziomie 0,1% (1)


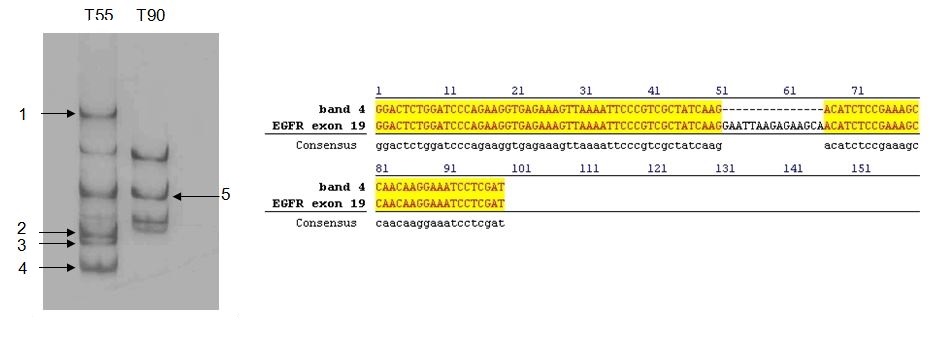
Delecja w ex.19 genu kodującego aktywną domenę wewnątrzkomórkowej kinazy EGFR jest genetycznym markerem lekooporności na terapie inhalatorami kinazy tyrozynowej (EGFR TKIs).
Panel lewy, u pacjenta T55 wykryto metodą MSSCP
obecność
zarówno wariantu zmutowanego (prążek nr 4) jak i wersję dziką (bez mutacji) - prążek nr 5. Ilościowe proporcje komórek pobranych w czasie biopsji oraz czułość wykrywania mniejszościowych wariantów genetycznych zastosowanej metody do genotypowania określają prawdopodobieństwo uzyskania fałszywie ujemnego wyniku w czasie analizy tego materiału.